
ggplotのgeom_signifを使って有意差を描きこんだグラフを作る。
データはRに組み込まれているサンプルデータであるirisのデータセットを利用します。
3群でLengthを比較
手順
- irisデータを読み込み
- Shapiro-Wilk testで正規性の検定
- ↑ p < 0.05ならノン・パラメトリック検定
- ggplotでグラフを書く
- geom_signifで有意差を書き込んでいく
- 最後にグラフの全体を調整して仕上げる
とりあえず、scriptの全貌
〜 help(iris) #description of iris data dat <- iris # import iris data attach(dat) library(dplyr) library(psych) # show overall summary describeBy(iris,Species) # show only length of each species describeBy(Sepal.Length ,Species) #Shapiro test if p<0.05 use non-parametric test shapiro_Sepal.Length <- shapiro.test(dat$Sepal.Length) shapiro_Sepal.Length #show p value #non parametric test p_value_Sepal.Length <- pairwise.wilcox.test(dat$Sepal.Length, df_length$Species) p_value_Sepal.Length #show p vaue ################################### #library for plot library(ggplot2) library(ggsignif) library(gridExtra) library(ggpubr) library(rstatix) p_length <- ggplot(dat , mapping = aes(x = Species, y = Sepal.Length)) p_length <- p_length + geom_boxplot(fill = "lightblue") p_length p_length <- p_length + geom_signif(comparisons = list(c("setosa", "versicolor")), map_signif_level=TRUE, y_position = 7, annotation="** p<0.001", fontface="italic", textsize=10) p_length <- p_length + geom_signif(comparisons = list(c("virginica", "versicolor")), map_signif_level=TRUE, y_position = 8, annotation="** p<0.001", fontface="italic", textsize=10) p_length <- p_length + geom_signif(comparisons = list(c("setosa", "virginica")), map_signif_level=TRUE, y_position = 8.8, annotation="** p<0.001", fontface="italic", textsize=10) # adjusting of y lim p_length <- p_length + ylim(2,10) # change to white back groud p_length <- p_length + theme_classic() # adjusting of aspect ratio p_length <- p_length + theme(aspect.ratio=1) # adjusting of aspect ratio p_length <- p_length + theme(text = element_text(size = 30)) p_length 〜
できあがりはこんな感じ
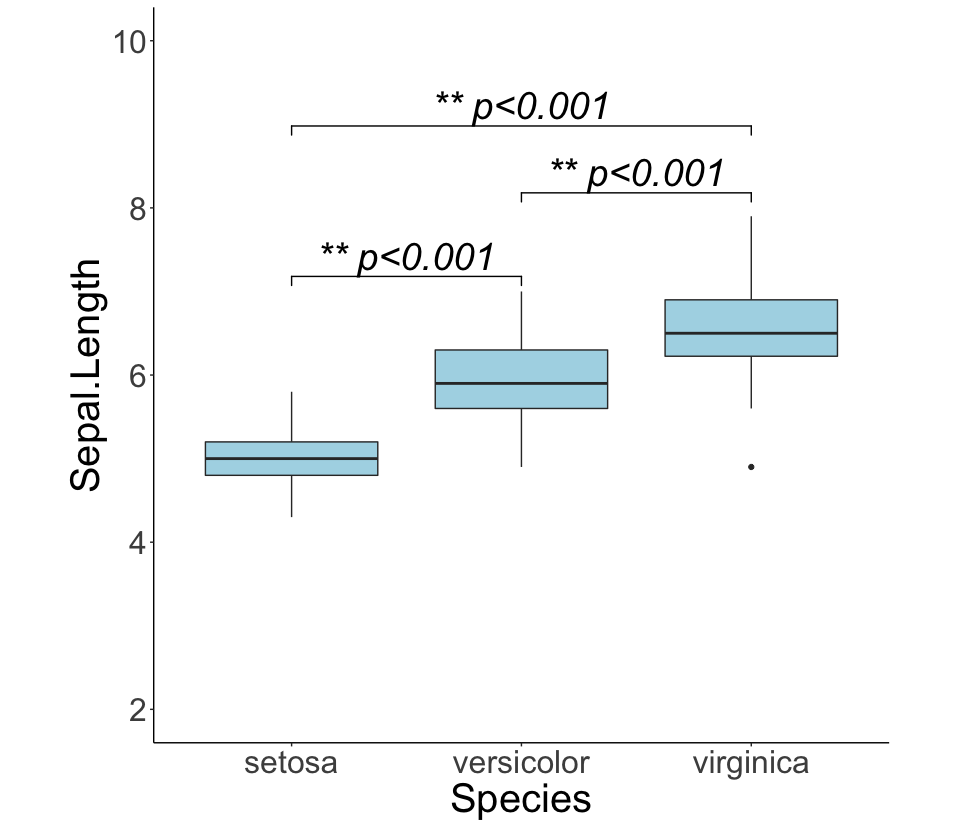
↓↓↓に各パールを解説
このように書くとirisのデータの内容を確認できる。
〜 dat <- iris # import iris data attach(dat) iris 〜
サクッと全体像を確認。
〜 # show overall summary describeBy(iris,Species) # show only length of each species describeBy(Sepal.Length ,Species) 〜

正規性を検定。p値を確認。今回はwilcoxonの検定を使う。
〜 #Shapiro test if p<0.05 use non-parametric test shapiro_Sepal.Length <- shapiro.test(dat$Sepal.Length) shapiro_Sepal.Length #show p value #non parametric test p_value_Sepal.Length <- pairwise.wilcox.test(dat$Sepal.Length, df_length$Species) p_value_Sepal.Length #show p vaue 〜
Shapiro testの結果

wilcoxonの結果

とりあえず、グラフをみてみる。
〜 p_length <- ggplot(dat , mapping = aes(x = Species, y = Sepal.Length)) p_length <- p_length + geom_boxplot(fill = "lightblue") p_length 〜

geom_signifを使って有意差の結果を入れていく。
そして、その後に微調整
〜 p_length <- p_length + geom_signif(comparisons = list(c("setosa", "versicolor")), map_signif_level=TRUE, y_position = 7, annotation="** p<0.001", fontface="italic", textsize=10) p_length <- p_length + geom_signif(comparisons = list(c("virginica", "versicolor")), map_signif_level=TRUE, y_position = 8, annotation="** p<0.001", fontface="italic", textsize=10) p_length <- p_length + geom_signif(comparisons = list(c("setosa", "virginica")), map_signif_level=TRUE, y_position = 8.8, annotation="** p<0.001", fontface="italic", textsize=10) # adjusting of y lim p_length <- p_length + ylim(2,10) # change to white back groud p_length <- p_length + theme_classic() # adjusting of aspect ratio p_length <- p_length + theme(aspect.ratio=1) # adjusting of aspect ratio p_length <- p_length + theme(text = element_text(size = 30)) p_length 〜
↑のできあがりのグラフになる。
今回は群間の比較をバラバラで行った。
scriptの書き方で一連で終える方法もあるようです。(群の組み合わせとp値をlist化する)
3群くらいであれば確認しながらできるので、この方法でもOKかと思います。